为深入理解复杂的生态系统,必须精确掌握生物种类及其丰度。对于肉眼可见的大型生物,通常能够通过直接观察进行计数。然而,对于大小仅为几微米且数量庞大的细菌,采用“计数DNA(基因)”的方法才是有效的。理化学研究所生命机能科学研究中心(理研BDR)细胞系统动态预测研究团队的负责人城口克之等人在《iMetaOmics》期刊上发表综述文章,概括了该技术领域的发展历程,通过介绍标记基因解析由多种细菌构成的“细菌群落”生态系统的技术,阐述了该技术对未来生命科学研究的潜在影响。
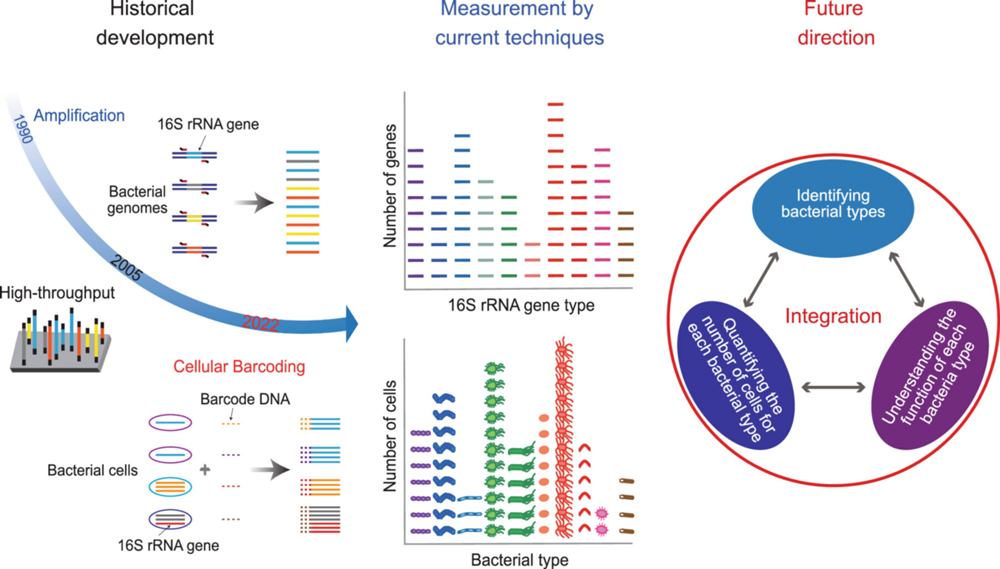
基于16S rRNA基因进行细菌物种鉴定和细胞数定量,并结合对各细菌功能的理解进行综合的细菌群落分析。(供图:理化学研究所生命机能科学研究中心、原图出自iMetaOmics刊登论文)
用于细菌群落解析的主要标记基因是16S核糖体RNA(16S rRNA)基因。这一区域的DNA序列在不同物种之间一般具有显著差异,因此成为鉴定细菌群落中存在细菌的重要线索。自1977年该方法首次被论证以来,已经经过了多次开发与改进。现在,这一分析方法已成为研究与人类健康密切相关的共生细菌群落的重要工具。
城口克之团队负责人等在2022年开发了一种新型细菌群落测量方法——“BarBIQ(Barcoding Bacteria for Identification and Quantification)法”注)。这种方法虽然与传统方法一样使用16S rRNA基因测序,但能够更准确地区分细菌,并且可以精确测量细菌的细胞数量。使用BarBIQ法,可以准确捕捉诸如饮食习惯变化引起的肠道细菌群落的微小变化。
关于细菌群落研究的未来发展,城口克之团队负责人指出:“目前还存在无法区分非常近缘的细菌种类的情况,所以还有进一步改进的空间”。然而,即便在现阶段,利用现有技术获取定量和定性数据仍具有重要意义。通过BarBIQ法,人类现在能够准确了解多种细菌的细胞数量和存在比例。如果将这些信息与每种细菌的全基因组解析所得到的基因和代谢通路等功能信息相结合,就能够更深入地理解整个细菌群落的行为。此外,通过整合基于细胞数测量的细菌分泌物分子量等其他数据,并进行数学分析,有可能准确预测因干预等因素引起的细菌群落变化。
生命科学的发展一直依赖于分析技术的进步以及对生命现象的理解。城口克之团队负责人在其综述中总结指出:“在细菌群落研究中,新的测量技术带来的变革期即将到来”。
文 理化学研究所生命机能科学研究中心 宣传部
注)参见2022年2月22日 客观日本 “理研等成功开发出高通量菌群分析新技术,开辟单细胞水平菌群测定的先河”
【论文信息】
期刊:iMetaOmics
论文:Long journey of 16S rRNA-amplicon sequencing toward cell-based
functional bacterial microbiota characterization.
DOI: doi.org/10.1002/imo2.9