东京大学生产技术研究所坪山幸太郎讲师(研究当时为美国西北大学博士后)和美国西北大学Gabriel Rocklin助教等人组成的研究团队成功构建了一种高效测量蛋白质结构稳定性的方法。蛋白质的结构稳定性是衡量蛋白质保持特定功能结构的指标,并定义了功能蛋白质分子的比例。以往的方法一次实验只能测量一种蛋白质的结构稳定性,但此次开发的新方法,一次实验可以测量约90万种蛋白质的结构稳定性。相关研究成果已发表在《Nature》上。
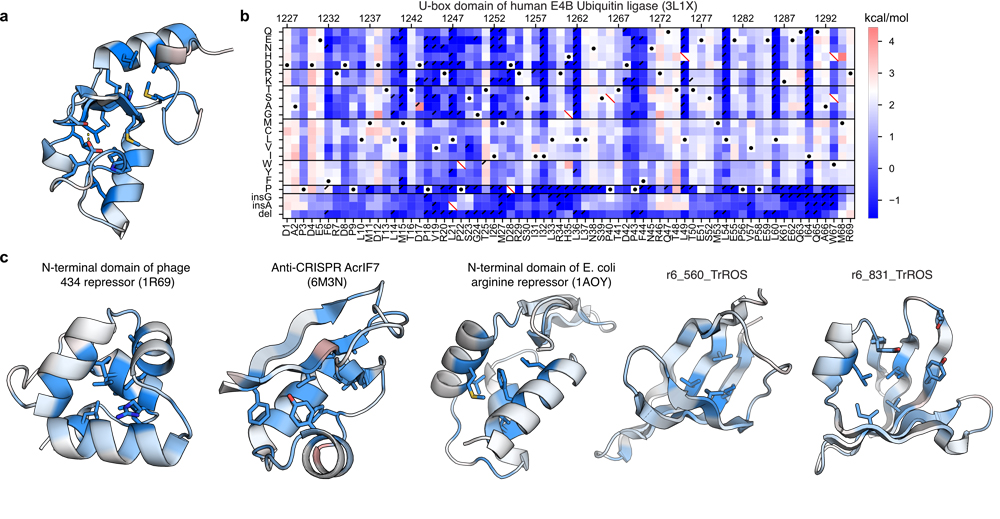
图1 蛋白质结构稳定性的解析图(供图:东京大学)
(a,c)显示了每个蛋白质位置对结构稳定性的重要性(蓝色越深越重要,红色越深越不重要)。(b)(a)蛋白质结构图的原始数据。与野生型蛋白质的结构稳定性(白、黑点)相比,结构稳定性更低(蓝)或结构稳定性更高(红)。为了制作单个蛋白质的“解剖图”,需要测量千种以上的氨基酸序列的结构稳定性。横轴代表蛋白质中的位置,纵轴代表氨基酸的置换、缺失和插入。
关于生物体内的蛋白质,人们熟知的是“氨基酸序列决定了蛋白质的结构,进而决定其功能”的安芬森法则。几乎所有蛋白质的功能都由其结构决定。此外,大多数蛋白质会在无结构的解体状态和整齐折叠的结构状态等多个状态之间转换。因此,显示蛋白质整齐折叠结构的分子比例的“蛋白质的结构稳定性”,即代表了功能性蛋白质的比例,是蛋白质最重要的特征数据之一。
事实上,当蛋白质的结构稳定性降低时,可能导致蛋白质之间发生非预期的相互作用,或者导致功能性蛋白质不足,从而引发癌症等各种疾病。
尽管如此重要,但正确理解蛋白质的结构稳定性,根据蛋白质的氨基酸序列和结构来预测蛋白质的结构稳定性是很困难的,需要耗费大量的心力、成本和时间。因此,即使整合了过去数十年来测定的蛋白质结构稳定性的数据库,也不过只包含了约3万种蛋白质的信息。此外,由于这些信息是从各种研究论文中整合而来,测定条件和数据质量各异,因此难以综合理解蛋白质的结构稳定性。
研究团队通过在两个方面下功夫,开发了一种高效测量蛋白质结构稳定性的方法。
第一个方面是,将蛋白质的氨基酸序列信息转换成DNA核酸信息。利用可以将蛋白质与相应的cDNA相结合的cDNA显示法,读取cDNA序列来确定氨基酸序列。通过结合cDNA显示法和次世代DNA测序技术两种技术,实现了一次解析大量类型的蛋白质氨基酸序列。
第二个方面是,为了量化蛋白质的结构稳定性,使用了蛋白质切割酶。蛋白酶可以切割已经解体的不稳定状态的蛋白质,结构稳定性较高的蛋白质可以被缓慢地切断,而结构稳定性较低的蛋白质会被迅速切割。也就是说,通过测量蛋白质被切割的速度,可以测量蛋白质的结构稳定性。
由此研究团队成功地在一次实验中测量了约90万种蛋白质的结构稳定性。研究人员多次重复该实验,从中选择高质量的数据,最终获得并公开了约80万种蛋白质结构稳定性的数据组。该数据库不仅规模庞大,而且具备在同一测量条件下测量的优势。
此外,研究人员还研究了约500种天然蛋白质和人工蛋白质,调查了各个蛋白质结构的保持机制。针对每个蛋白质,分别对每个位置的氨基酸进行了20种不同的氨基酸置换和缺失,并插入甘氨酸或丙氨酸测量了其结构稳定性。以此实现了对每个蛋白质中哪些部位对维持结构稳定性至关重要等结构稳定性的可视化。
同时,研究人员还调查了生物体内蛋白质中各种氨基酸的易用性与氨基酸带来的结构稳定性之间的关系。发现越是能稳定结构的氨基酸越容易被使用,并且能够量化这种趋势。此外,还研究了排除结构稳定性的影响时氨基酸的使用情况,结果显示,易溶于水的亲水性氨基酸更容易被使用,而不易溶于水的疏水性氨基酸,尤其是含有芳香环的芳香族氨基酸,使用频率较低。
由于该数据库与以往相比规模庞大,因此有可能揭示蛋白质结构稳定性的重要性质以及与结构稳定性相关的普遍规律。此外,大规模的结构稳定性数据将成为蛋白质科学AI开发的基础。有望通过这种AI帮助识别导致疾病的氨基酸突变或更有效地合成蛋白质药物。
原文:《科学新闻》
翻译:JST客观日本编辑部
【论文信息】
杂志:Nature
论文:Mega-scale experimental analysis of protein folding stability in biology and design
DOI:10.1038/s41586-023-06328-6