日本理化学研究所(简称“理研”)生命机能科学研究中心计算分子设计研究团队的小松辉久研究员、小山洋平研究员、冲本宪明高级研究员、森本元太郎技师、大野洋介高级技师和泰地真弘人组长组成的研究团队,利用分子动力学(MD)模拟专用计算机“MDGRAPE-4A”,对新型冠状病毒感染症(COVID-19)的致病病毒“SARS-CoV-2”的主蛋白酶结构动态进行了10微秒(1微秒为100万分之1秒)的模拟。并于3月17日在存储库Mendeley Data中公开了原始数据,以供全球的新药研究人员自由使用。
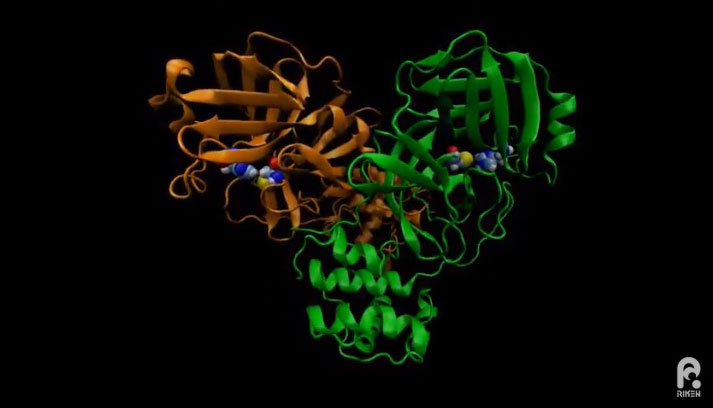
图:MDGRAPE-4A超算模拟的SARS-CoV-2主蛋白酶结构。用颜色区分了构成二聚体的两个亚基,用球形模型表示了位于蛋白酶活性中心的氨基酸位点。(理研公开视频截图)
该数据有望用来开发有效抑制病毒繁殖所必需的蛋白酶活性的抑制剂,以及筛选候选分子等。
冠状病毒的基因组为单链RNA,感染宿主细胞后,会通过RNA基因组翻译出长蛋白(多聚蛋白)。随着这种多聚蛋白被裂解,各片段会作为病毒繁殖所需的结构蛋白和酶发挥作用。在这些酶中,主要催化多聚蛋白裂解的是主蛋白酶。抑制主蛋白酶裂解活性的抗病毒药物正在开发之中,已有多家研究机构报告了针对人类免疫缺陷病毒(HIV)等现有病毒的蛋白酶抑制剂和SARS-CoV-2主蛋白酶复合体的立体结构(X射线晶体解析数据)。MD模拟是在近似于细胞内的环境下解析分子间相互作用的手法,可用来获取高精度推进新药筛选(考虑水溶液中的分子动态和溶剂效果)的信息。
研究团队以最近报告的SARS-CoV-2主蛋白酶X射线晶体解析数据为基础,利用MDGRAPE-4A模拟了蛋白酶二聚体在水溶液中的结构动态。
视频:利用MDGRAPE-4A模拟的SARS-CoV-2的主蛋白酶结构动态(理研公开视频)
研究背景
新型冠状病毒感染症(COVID-19)自2019年在中国武汉首次报告病例以来,至今已扩散到世界各地,世界卫生组织(WHO)宣布疫情进入全球大流行。这种新型病毒产生的社会影响已波及到全球,开发针对COVID-19的有效药物成为非常紧急的国际事件。
冠状病毒的基因组为单链RNA,感染宿主细胞后,会通过RNA基因组翻译出长蛋白(多聚蛋白)。随着这种多聚蛋白被裂解,各片段会作为病毒繁殖所需的结构蛋白和酶发挥作用。确定COVID-19的致病病毒为“SARS-CoV-2”后,很多研究机构都解析了病毒蛋白的立体结构,并不断将成果录入公共数据库。
根据结构解析开发抗病毒药物时,靶蛋白之一是主要催化多聚蛋白裂解的主蛋白酶。抑制主蛋白酶的裂解活性被认为是可以抑制病毒在感染细胞内的繁殖,所以科学家都在寻找与主蛋白酶结合的抑制性分子。实际上,现在已针对人类免疫缺陷病毒(HIV)开发出了多种靶向主蛋白酶药物,并显示出了有效性。
在近年来的药品开发中,利用超级计算机探索和评估针对靶蛋白的候选药物分子的计算机模拟(In Silico)新药开发备受关注。分子动力学(MD)模拟是在近似于细胞内的环境下解析分子间相互作用的方法,可获得用来高精度推进新药筛选(考虑水溶液中的分子动态和溶剂效果)的信息。
研究团队开发了新药开发专用超级计算机“MDGRAPE-4A”,从2019年11月开始实验性运行。与普通计算机相比,MDGRAPE-4A擅长在短时间内模拟蛋白质的结构变化,因此对靶蛋白、候选药物分子和水分子的动态结构进行采样后,有望利用其结构数据进行高精度的结合亲和性预测,并以现实的计算时间实现高精度的计算机模拟筛选。
研究方法与成果
研究团队利用MDGRAPE-4A超算,以可以自由利用的形式公开了SARS-CoV-2主蛋白酶的MD模拟数据。用于模拟的SARS-CoV-2主蛋白酶的立体结构主要以Liu等人的报告为基础,溶剂的水分子数量将近3万个,系统的总原子数量将近10万个。利用MDGRAPE-4A实施了约10天的计算,模拟了SARS-CoV-2主蛋白酶的10微秒(1微秒为100万分之1秒)结构动态(图1)。
一般认为,水溶液中的蛋白质与药物的分子间相互作用发生在亚毫秒(~100微秒)的时间尺度内,此次的数据应该可以作为推算药物分子结合前的主蛋白酶结构动态的基础数据。
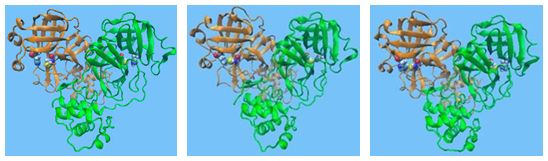
图1:利用MDGRAPE-4A模拟的SARS-CoV-2的主蛋白酶结构动态。从图中可以看出,主蛋白酶结构会随着时间而产生变化。
未来展望
目前,作为候选的抗病毒药物,已提出了几种能够与SARS-CoV-2主蛋白酶结合的分子,而且对其复合体实施了X射线晶体解析。虽然距离真正开发出新药还有很长的路要走,还需要开展大量的研究,但利用此次公开的MD模拟数据可预测候选分子与主蛋白酶的结合强度等,有望加快国内外的新药研究速度。
今后,研究团队预定继续模拟SARS-CoV-2的主蛋白酶,并广泛研究结合状态下的候选药物分子的模拟、类似蛋白酶,以及在SARS-CoV-2中编码的其他潜在新药靶蛋白的模拟等。
文:JST客观日本编辑部编译