日本大学共同利用机关法人 自然科学研究机构分子科学研究所(以下简称“分子研”)的原岛崇德助教、大友章裕助教、饭野亮太教授等组成的研究团队发表科研成果称,通过对人工分子马达的一种——DNA纳米粒子马达的运动基元的反应过程与化学反应基元过程进行解析,成功提升了人工分子马达的运动性能,同时实现了30纳米/秒的运动速度与3微米的行进距离,其运动性能与天然马达蛋白相当。借助这项突破,未来有望打造出通过人工分子马达运动进行计算的分子计算机,以及通过人工分子马达运动高灵敏度检测感染与疾病分子的检测系统。相关研究成果已于1月16日发表在国际学术期刊《Nature Communications》的网络版上。
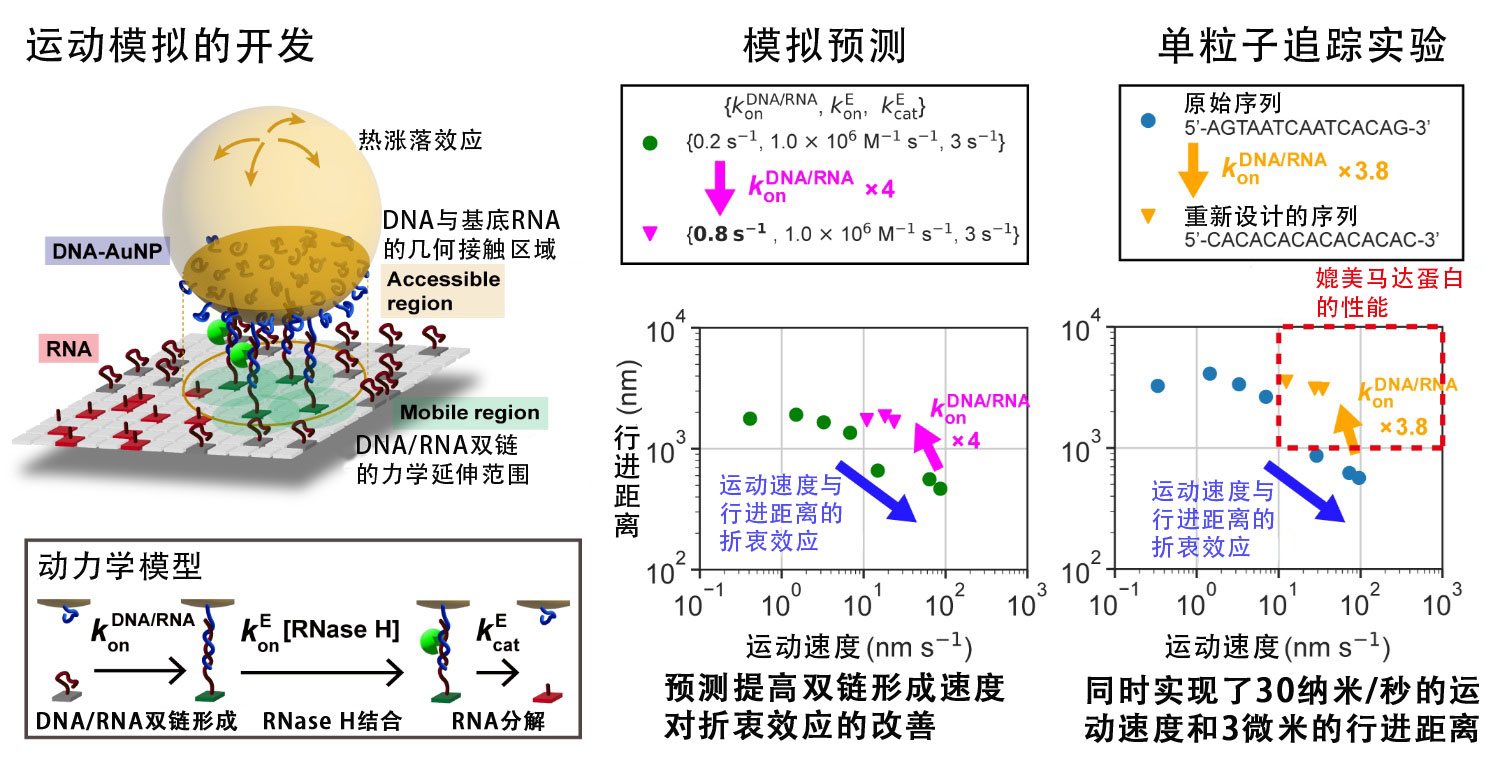
图1:基于模拟预测,重新设计的DNA碱基序列,成功实现了性能提升(供图:分子科学研究所)
已知天然马达蛋白如驱动蛋白和肌球蛋白是以ATP(三磷酸腺苷)分子作为能量来源的,能够实现10-1000纳米/秒的速度,在细胞内的物质运输和肌肉收缩等生命活动中发挥着重要作用。
学术界正在着手人工设计分子马达,并探索将其用于纳米级运输器件等领域的研究。其中,利用DNA分子构建的DNA纳米粒子人工马达,因碱基序列与结构的高度可设计性而被认为极具发展潜力。然而,以往的DNA人工分子马达存在运动速度较慢的问题,仅为0.1纳米/秒。即便是经金纳米粒子修饰后DNA的人工马达,运动速度最快也只有1纳米/秒左右,需要提升运动性能。
此次,研究团队的目标是明确并优化DNA纳米粒子马达(由各向同性球形金纳米粒子构成)的运动瓶颈过程。
通过高速高精度单粒子追踪实验,研究团队发现,DNA纳米粒子马达并非以恒定速度连续运动,而是像马达蛋白一样,通过暂停(pause)与瞬间移动(step)交替进行的“pause-step”运动模式来移动。
研究还发现,运动速度较慢的原因在于其暂停时间长达70秒,比马达蛋白要长。
运动暂停期间,化学反应基元过程继续进行,其中的RNase H(核糖核酸酶H)结合过程被确认为关键瓶颈步骤。
研究人员通过加速这一结合过程后发现,暂停时间虽然缩短到了0.2秒左右,但会出现行进距离缩短的折衷效应。
为了查明其原因,研究团队开发出一款运动再现模拟系统,并基于“若将DNA/RNA双链形成速度提升至原来的4倍,则可以在不发生折衷效应的情况下兼顾运动速度与行进距离”的预测设计出了DNA纳米粒子马达。
结果表明,研究团队将双链形成的速度提高了3.8倍。
当使用这种序列设计出的马达进行实验后发现,它与天然马达蛋白相媲美,并同时实现了30纳米/秒的运动速度与3微米的行进距离。
原岛助教表示:“DNA不仅是掌控遗传信息的生物分子,同时也被认为是构建人工分子马达等纳米材料的重要组成部分。通过推动这项研究,我们或许可以实现即便在肉眼不可见的纳米世界中,也能精确运输物质的传输系统,以及利用运动进行计算的分子计算技术等广泛应用。”
原文:《科学新闻》
翻译:JST客观日本编辑部
【论文信息】
期刊:Nature Communications
论文:Rational engineering of DNA-nanoparticle motor with high speed and processivity comparable to motor proteins
DOI:10.1038/s41467-025-56036-0