东京大学研究生院农学生命科学研究科岩田洋佳教授等通过整合以及聚类分析植物根际土壤或动物肠道等宿主生物周边的微生物组(Microbiome)与代谢物组(Metabolome)的数据,开发出了能够明确微生物与代谢物关系的“整合概率变分变量选择”(I-SVVS)新方法。该方法有望深化理解多种宿主生物中微生物组与代谢物组的相互作用,推动农业、医疗及环境科学发展。相关研究成果已发表在《Briefings in Bioinformatics》上。
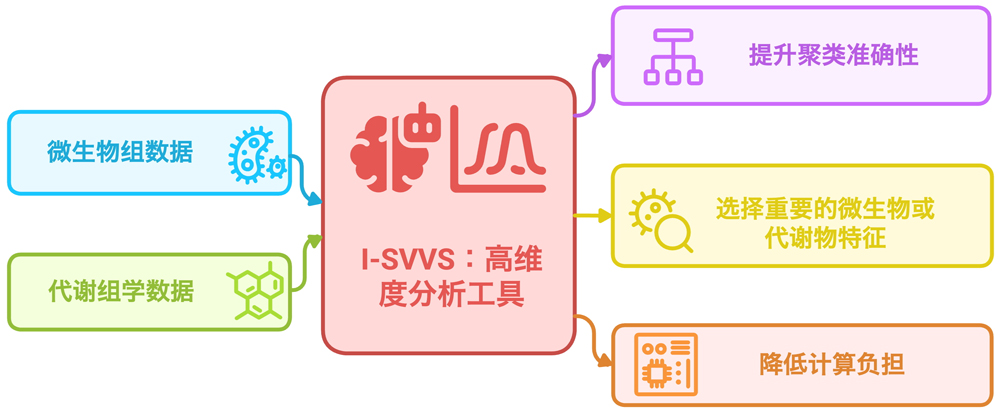
图1 用于多组学微生物组研究的I-SVVS解析法(供图:东京大学)
多组学微生物组数据是指用于理解微生物群落功能及相互作用,整合宏基因组(表征微生物种类的遗传信息)以及代谢组等各类组学技术的统合数据。这类数据具有信息量大,但数据点密度低、多个变量强相关(多重共线性)的特点,需要高级计算方法才能准确解析。
研究团队开发的I-SVVS法是首个专用于高维微生物组数据分析的方法。I-SVVS法采用基于概率变分推断的变量选择框架,对微生物数据应用无限狄利克雷多项分布混合模型,对代谢物数据应用无限高斯混合模型,从而提升计算效率与解析精度。此外,该方法能从数据内的海量特征中识别出包含关键信息的最小特征集,实现高精度分类(聚类)并发现微生物与代谢物的关联性。
使用大豆、小鼠和人类三组数据集进行的实验结果表明,I-SVVS在所有案例中均展现出比既往解析方法(iClusterPlus和Clusternomics)更高的精度与更快的处理速度。特别是在大豆数据集(377个样本、16,943种微生物、265种代谢物)中表现出更快速精准的解析能力。成功鉴定出与大豆生长相关的关键微生物群,并阐明了不同环境条件下微生物与代谢物的关联性。
上述结果表明I-SVVS有望大幅推动多组学微生物组研究。此前,难以明确微生物功能与宿主健康、生长的关系,解析复杂数据需耗费大量时间和精力。而使用I-SVVS法能更准确理解微生物如何与宿主生物相互作用,如何促进生长或引发疾病。
例如,该方法可详细解析土壤微生物如何促进植物生长,或肠道菌群如何影响人类健康。这在农业领域将有助于提高作物产量、推动可持续农业发展,在医疗领域可促进基于肠道菌群的疾病预防与治疗方法的开发。在环境领域也能深化理解微生物对生态系统平衡的作用,为自然环境保护和应对气候变化做出贡献。
未来通过利用I-SVVS法,有望进一步阐明微生物与宿主之间的关系,并在农业、医疗、环境科学等广泛领域实现应用。
原文:《科学新闻》
翻译:JST客观日本编辑部
【论文信息】
期刊:Briefings in Bioinformatics
论文:I-SVVS: Integrative stochastic variational variable selection to explore joint patterns of multi-omics microbiome data
DOI:10.1093/bib/bbaf132