要想加速萝卜品种的开发,需要完善基因组信息。日本东北大学研究生院农学研究科2014年全球率先发布了萝卜基因组碱基序列信息,为进一步增加基因组信息,此次东北大学又带头与千叶县Kazusa DNA研究所和农业食品产业技术综合研究机构(以下简称“农研机构”)蔬菜花卉研究部门合作,以当地品种为主,分析了全球各地500多种萝卜的DNA,明确了全球品种间的差异(SNP:单核苷酸多态性)。
根据遗传信息,全球的萝卜主要分为四大类,日本的品种单独为一个类别。详细研究内容预定发布在国际科学期刊《DNA Research》上,已于2020年3月13日率先发布于网络版。
利用此次的成果,控制萝卜特有特征(性状)的基因探索、利用基因组信息的育种(品种开发),以及识别品种的DNA分析技术开发均有望加速。
萝卜是全球广泛种植的十字花科蔬菜。欧洲等地区主要种植樱桃萝卜,南亚和东南亚种植食用种荚的鼠尾萝卜,东亚地区则种植根茎又大又粗的萝卜。日本各地都有自己独特的地方品种,目前已知的地方品种总共达100多种,除了普通的青头萝卜和白萝卜外,还有根茎为圆形的“圣护院萝卜”、直径达30cm的“樱岛萝卜”、形状细长达到2m以上的“守口萝卜”、比较辛辣的“辛味萝卜”,以及根茎较细、主要食用叶子的“小濑菜萝卜”等。
不过,此前对遗传上如何决定萝卜的这些特征知之甚少。2014年,以东北大学研究生院农学研究科的西尾刚教授(现为名誉教授)和北柴大泰副教授(现为教授)为中心组成的研究团队,全球率先发布了青头萝卜的基因组碱基序列信息,利用基因组信息的遗传学研究由此拉开帷幕。之后又有多家研究机构公开精度更高的基因组碱基序列信息。
但除青头萝卜以外,其他品种的基因信息无论是品种和品系数量还是信息量都非常少。要想找出决定形状和成分等差异的基因和决定不同品种特征的基因,需要通过大范围的染色体(全基因组)明确在很多萝卜品种和品系之间存在差异的庞大SNP(单核苷酸多态性)。近年来,随着新一代测序仪的出现和利用测序仪的分析技术的发展,已经可以迅速明确基因组信息,因此研究团队以全球的萝卜为对象,实施了全基因组SNP分析。
研究成果
1.以世界各地种植的萝卜(Raphanus sativus L.)的品种、野生品系,以及与R. sativus为近缘的其他野生品种(R. raphanistrum和R.maritimus)共约500种为对象,明确了染色体上约53,000个位置的SNP(图1)。分析的萝卜品种和品系数量为史上最多,检测出的总SNP数量约为2,100万,也是全球最多的。
图1:检测出的SNP在染色体上的分布
用红色显示了约53,000处SNP中的一部分的位置。Rs1~Rs9表示染色体编号。
2.整理这些数量庞大的信息,以研究人员和品种开发人员方便利用的形式构建了数据库。预定近日在Kazusa DNA研究所运营的网站http://radish.kazusa.or.jp上公开。
3.根据此次研究明确的遗传信息进行品系解析,确认了以下两点。
1)约500个品种和品系根据地理分布主要分为四类。日本的品种和品系与东亚(中国和韩国)不同,形成了独自的类别(图2)。
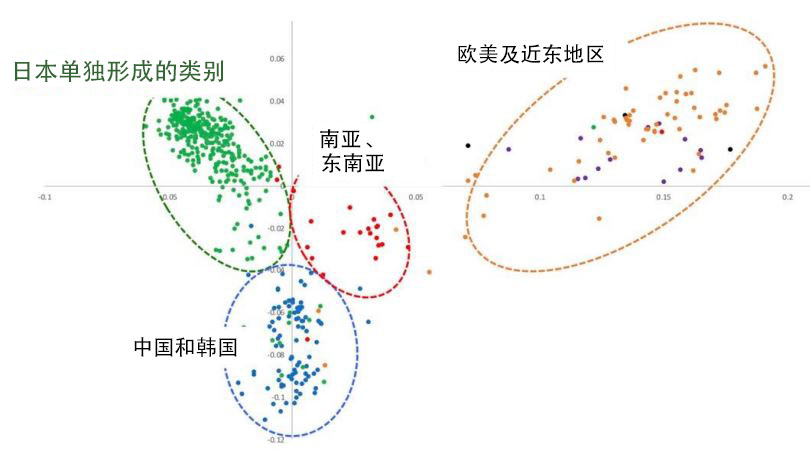
图2:根据SNP信息分析的各品种的遗传分布(主座标分析)
各图分别显示了品种,在计算的遗传位置绘制。绿色表示源自日本的品种和品系,形成了一个单独的类别。
2)日本的萝卜以前一直被认为是经由中国和韩国传入的。但此次解析发现可能存在新的传播途径,即经由南亚和东南亚传入西南诸岛和南九州,然后传播到日本国内。
未来展望
1. 萝卜的根茎形状和大小多种多样,不同品种的根茎颜色(白色、红色、紫色、黑色等)和辛辣程度也不同。除根茎以外还有食用种荚的萝卜,既有种荚很小,种子数量很少的类型,也有种荚长达20cm以上的萝卜。此外还有根茎较细,食用叶子的叶萝卜。有播种后很快(30天内)就开花的品种,也有播种后200多天才开花的品种。不同的品种和品系各有自己的特征。
利用此次的研究成果,有望加速探索控制这些特征(性状)的基因。另外,还有助于加速利用基因组信息的育种(品种开发)。
2. 此次研究明确了大量品种的庞大数量SNP,由此可提供用来对地方品种等进行品种间识别和品种鉴定的基因信息。今后有望促进使用SNP信息的识别和鉴定分析技术的开发。
3. 据说日本的地方品种超过100种。利用此次的SNP信息,可以明确地方品种的详细亲缘关系。另外,明确了独特性的地方品种作为遗传资源的重要性也将增加。应该可以利用这项成果的信息创建遗传资源保护指标。
4. 受《生物多样性公约》的《名古屋议定书》限制,现在很难获得海外各国和地区的遗传资源。因此,各国和地区通过以与此次研究相同的方式分析当地自古以来就有的萝卜品种和品系,并结合本次成果的数据进行解析,能更准确地查清全球萝卜的传播路径。
题目:Identification of genome-wide single-nucleotide polymorphisms among geographically diverse radish accessions
期刊:《DNA Research》
DOI:https://doi.org/10.1093/dnares/dsaa001
日语发表原文
文:JST客观日本编辑部